Что такое разварки в автомобиле? Как сделать своими руками?
Автомобилист Без рубрики Оставить комментарий
Для многих еще представляется диким тот факт, чтобы своими руками занижать автомобиль. Автомобили с искусственно заниженной посадкой теряют свои определенные способности по безопасности. Для авто с обычным клиренсом и искусственно заниженных машин начали ставить разварки. Разварки — это переделанные заводские колесные диски с целью изменения его размеров.
Содержание статьи:
- Как делают разварки?
- Ширина диска.
- Как сделать разварки своими руками?
- Плюсы и минусы.
- Видео.
Как делают разварки
Разварки — это диски с измененной шириной. Обычные заводские диски разрезают и сваривают. Для того, чтобы сделать один широкий диск, надо разрезать два диска, то из двух сделать один. Некоторые не портят второй диск, а наращивают на разрезанном диске ширину путем приварки стальной полосы.
Обычные заводские диски разрезают и сваривают. Для того, чтобы сделать один широкий диск, надо разрезать два диска, то из двух сделать один. Некоторые не портят второй диск, а наращивают на разрезанном диске ширину путем приварки стальной полосы.
Что дают широкие диски, для чего их делают и устанавливают? Ответ: для того, чтобы можно было поставить широкую резину, для того, чтобы диски выпирали из колесных арок, как бы для красоты, такой внешний тюнинг. Это удел нестандартной моды, в основном, этим занимается молодежь.
Увеличенные по ширине колесные диски не улучшают технические характеристики автомобилей, какой марки и модели ни были, а наоборот, аэродинамика ухудшается, управляемость незначительно ухудшается и увеличивается расход топлива.
Из каких дисков делают разварки
Дешевле и легче делать разварки из штампованных дисков, то есть те, которые устанавливают на заводе-изготовителе, самые простые. Литые и кованные диски непригодны для разрезания и сварки.
Литые или кованые диски нестандартной ширины делаются на заказ, для этого нужно оборудование и навыки. Стоимость таких дисков будет раза в два или в три дороже.
Ширина диска
Размеры обычного стандартного штампованного диска имеет размер 6,5 J. Цифры 6,5 — это ширина в дюймах (один дюйм равен 2,54 сантиметра, получается 6,5 х 2,54 = 16,51 см). Буква J (Джей) означает маркировку профиля боковой закраины обода. Существуют маркировки JJ, JK, K, L.
Так как большинство дисков имеют размер 6,5 J, букву некоторые не пишут, а просто обозначают ширину.
На такие диски можно поставить резину только стандартной такой же ширины. Если хочется протюнинговать колеса, чтобы они выделялись из колесных арок крыльев, например, поставить резину 10 J, то есть ширина больше на 3,5 дюйма, а это почти 9 см, что делать? Только путем разварки дисков.
Прежде чем разваривать диски, надо узнать, если ли на такие диски резина. Если нет, зачем их делать.
Разварки своими руками
Существуют три способа, как сделать разварки своими руками:
- Можно сделать широкий диск из двух обычных. Только для этого диски надо разрезать не по середине. Затем две части разных дисков, которые пошире, сварить между собой.
- Разрезать один диск по середине и вставить стальную пластину, соответствующую характеристикам металла диска и приварить все три части.
- Комбинированный способ труднее всего. Он заключается в разделении внутренней и наружной части диска, приваркой стальной полосы и перенесения ступичной части.
Как вы уже поняли, чтобы сделать разварку своими руками, необходимо обладать навыками сварщика, проварить нужно качественно.
А вот, чтобы наоборот приподнять автомобиль, для этого нужно установить специальные автобаферы Power Guard — здесь сейчас продаются с большой скидкой.
Плюсы и минусы
Плюсы и минусы, достоинства и недостатки есть всегда, вопрос, чего больше, а чего меньше.
К плюсам разварок можно отнести:
- Необычный вид колес автомобиля. Раньше старались купить подороже с оригинальным рисунком литье или кованные, теперь перешли на изменение ширины колес.
- Недорогой внешний тюнинг.
- Лучшее сцепление с дорожным покрытием, площадь больше.
К минусам можно отнести:
- Стандартные колеса марки R13/R14, чтобы были широкие, невозможно.
- Цельный диск есть цельный, а приваренный, как бы качественно он ни был приварен, имеется вероятность, что сварной шов развалится.
- Когда придется менять камеры или, если это бескамерка, клеить резину, шиномонтажнику с балансировкой колес придется повозиться.
- Скорость автомобиля и ускорение становятся меньше, так как колеса стали тяжелее.
- Расход топлива вырастает до 20%.
- Подшипники ступицы работают с повышенными нагрузками, потому что они не рассчитаны для дисков такой ширины.
- Зимой не попадает в колею, а это опасно.
Недостатков данного улучшения внешнего вида авто больше. Да и улучшение ли это? Пишите в комментариях свои мнения.
Да и улучшение ли это? Пишите в комментариях свои мнения.
Видео
Что такое разварки?
Пять причин не ставить разварки.
Автор публикации
Как варить рис, чтобы он получился идеальным
Рассказываем, как приготовить крупу в кастрюле, сковороде, мультиварке и микроволновке.
Как подготовить рис
Если вы хотите сварить рассыпчатый рис, перед приготовлением его нужно промыть под холодной водой. Так вы избавитесь от крахмала, который отвечает за клейкость. Промойте рис около пяти раз или больше, пока вода не станет прозрачной. Удобнее всего выполнять эту процедуру с помощью мелкого сита.
Кадры: Tasty / YouTubeДля приготовления некоторых блюд, таких как ризотто, нужен клейкий рис. В этом случае промывать его не стоит. Либо можно ограничиться одним ополаскиванием, чтобы смыть всё лишнее.
Чтобы рис сварился быстрее, его можно замочить на 30–60 минут. Тогда время приготовления сократится почти вдвое. Однако при этом лучше уменьшить количество воды, которое используется для варки.
Тогда время приготовления сократится почти вдвое. Однако при этом лучше уменьшить количество воды, которое используется для варки.
Сколько воды взять для варки риса
Обычно считается, что для приготовления этой крупы нужно в два раза больше воды. Но это приблизительная пропорция. Лучше отмерять объём жидкости исходя из вида риса:
- для длиннозёрного — 1 : 1,5–2;
- для среднезёрного — 1 : 2–2,5;
- для круглозёрного — 1 : 2,5–3;
- для пропаренного — 1 : 2;
- для коричневого — 1 : 2,5–3;
- для дикого — 1 : 3,5.
Обязательно читайте инструкцию на упаковке. Производитель точно знает, какой обработке подвергся рис, и подсказывает оптимальный для крупы объём воды.
Отмеряйте рис и воду мерным стаканом — так гораздо удобнее. Стандартная порция на одного — 65 мл сухих зёрен.
Что добавить к рису
Вам точно понадобится соль. На 1 стакан зёрен достаточно ¹⁄₂ чайной ложки. Хотя лучше регулировать количество по своим предпочтениям.
Рис хорош тем, что его вкус всегда можно немного изменить. Например, с помощью следующих приправ:
- шафран;
- карри;
- кардамон;
- зира;
- тмин;
- корица;
- гвоздика.
Специи добавляются в воду при варке или в уже готовое блюдо.
Также рис можно дополнить пряными травами, цедрой цитрусовых или сварить не на воде, а на мясном или курином бульоне.
Сколько варить рис
Это зависит от способа приготовления и вида риса. Поэтому время варки может варьироваться от 20 до 60 минут. Ниже вы найдёте все подробности.
Как варить рис в кастрюле
Лучше использовать посуду с толстым дном: в ней температура распределяется равномерно.
Сначала доведите подсоленную воду до кипения, а после высыпьте в неё крупу. Перемешайте рис один раз, чтобы зёрнышки не прилипали ко дну. Затем дождитесь, когда блюдо начнёт бурлить, убавьте огонь до минимума и накройте кастрюлю крышкой.
Кадры: Downshiftology / YouTubeВ процессе приготовления не поднимайте крышку — иначе рис будет готовиться дольше. Если вы хотите, чтобы крупа была рассыпчатой, не перемешивайте её (за исключением первого раза). В противном случае зёрна будут ломаться и выделять крахмал.
Если вы хотите, чтобы крупа была рассыпчатой, не перемешивайте её (за исключением первого раза). В противном случае зёрна будут ломаться и выделять крахмал.
Среднее время варки после повторного закипания в зависимости от вида риса составляет:
- для белого — 20 минут;
- для пропаренного — 30 минут;
- для коричневого — 40 минут;
- для дикого — 40–60 минут.
Когда рис приготовится, снимите его с огня и дайте постоять 10–15 минут под крышкой. Если в готовой каше осталась вода, слейте её или накройте кастрюлю сухим полотенцем: оно впитает излишки влаги.
Кадры: Downshiftology / YouTubeКак варить рис в сковороде
Используйте сковороду диаметром от 24 см, высокими бортиками и крышкой.
Рис готовится в ней почти так же, как в кастрюле, за исключением одного нюанса: зёрна предварительно нужно быстро обжарить на растительном масле. Делайте это 1–2 минуты, постоянно помешивая, чтобы зёрнышки покрылись маслом, тогда рис будет рассыпчатым. Затем его нужно залить кипятком и готовить так же, как и в способе выше.
Затем его нужно залить кипятком и готовить так же, как и в способе выше.
Как варить рис в мультиварке
Выложите рис в чашу мультиварки. Добавьте воду и нужные вам специи. Закройте крышку и установите режим «Крупы», «Рис», «Плов» или «Гречка». Примерное время варки риса составляет:
- для белого — 30 минут;
- для пропаренного — 30–40 минут;
- для коричневого — 50 минут;
- для дикого — 50–60 минут.
Как варить рис в микроволновке
Высыпьте рис в ёмкость, пригодную для микроволновки. Желательно, чтобы крупа не занимала больше ¹⁄₃ объёма посуды. Добавьте воду и специи.
Белый и пропаренный рис готовьте 15–20 минут на полной мощности. Затем перемешайте, накройте крышкой и оставьте в закрытой выключенной микроволновке на 5–10 минут.
Коричневый и дикий рис сперва варите 5 минут на полной мощности. Затем перемешайте, установите среднюю мощность и готовьте ещё 20–25 минут. Оставьте под крышкой в закрытой микроволновке на 10–15 минут.
Если зёрна остались твёрдыми, продолжайте варить, проверяя каждые 1–2 минуты и доливая воду, если это необходимо.
Бонус: как приготовить рис для суши
- Для приготовления суши используется специальный японский рис. Заменить его можно обычным круглозёрным.
- Перед приготовлением крупу нужно промыть 5–7 раз. Всплывшие зёрна лучше выбросить.
- Промытый рис залейте холодной водой в пропорции 1 : 1,5. В кастрюлю можно добавить кусочек водорослей нори для вкуса, но перед закипанием их нужно удалить.
- Рис варится под крышкой: до закипания — на среднем огне, после — на минимальном около 15 минут. После нужно снять крупу с плиты и дать ей постоять ещё 15 минут.
- Готовый рис необходимо приправить специальной заправкой. Для её приготовления влейте в отдельную кастрюлю 2 столовые ложки рисового уксуса, добавьте 1 чайную ложку сахара и 1 чайную ложку соли и подогрейте смесь на среднем огне до полного растворения сыпучих ингредиентов.
- Переложите рис в широкую миску, полейте соусом и осторожно перемешайте деревянной лопаткой.
 После этого остудите и приступайте к приготовлению суши.
После этого остудите и приступайте к приготовлению суши.
Этот материал впервые был опубликован в январе 2017 года. В сентябре 2020-го мы обновили текст.
Читайте также 👩🍳🥣👌
- Рисовая каша с фрикадельками
- Рисовый бургер с креветками темпура
- Лечо с рисом на зиму
- Помидоры, фаршированные рисом и овощами
- Суп с лисичками и рисом
мРНК — Biology LibreTexts
- Последнее обновление
- Сохранить как PDF
- Идентификатор страницы
- 23915
- Кэтрин Харрис
- Колледж Хартнелл
Транскрипция и процессинг (который включает сплайсинг) новообразованной мРНК происходит в ядре клетки.
После образования зрелого транскрипта мРНК его транспортируют в цитоплазму для трансляции в белок.
Большинство эукариотических генов и их транскриптов пре-мРНК содержат некодирующие участки нуклеотидов или области, которые не предназначены для превращения в белок. Эти некодирующие сегменты называются интронов и должны быть удалены до того, как зрелая мРНК сможет транспортироваться в цитоплазму и транслироваться в белок. Участки ДНК, кодирующие аминокислоты в белке, называются экзонами . В процессе сплайсинга интроны удаляются из пре-мРНК сплайсосомой, а экзоны снова соединяются вместе. Если интроны не удалить, РНК будет транслироваться в нефункциональный белок. Сплайсинг происходит в ядре до того, как РНК мигрирует в цитоплазму. После завершения сплайсинга зрелая мРНК (содержащая непрерывную кодирующую информацию) транспортируется в цитоплазму, где рибосомы переводят мРНК в белок.
Подробный обзор сплайсинга мРНК
Транскрипт пре-мРНК
Транскрипт пре-мРНК содержит как интроны, так и экзоны. Интроны удаляются в процессе сплайсинга. В этом примере пре-мРНК содержит два экзона и один интрон.
Рисунок \(\PageIndex{2}\). (CC BY-NC-SA; Agathman )
Интроны содержат несколько важных и консервативных последовательностей, управляющих процессом сплайсинга: 5’-последовательность GU (
)0040 5’-сайт сплайсинга ), сайт разветвления A , расположенный рядом с богатой пиримидином областью (область с большим количеством цитозиновых и урациловых оснований), и 3’-последовательность AG (3’-сайт сплайсинга ).
Рисунок \(\PageIndex{3}\). (CC BY-NC-SA; Agathman )
Сплайсосома
Большой белковый комплекс, известный как сплайсосома , контролирует сплайсинг мРНК. Сплайсосома состоит из частиц, состоящих как из РНК, так и из белка.
Сплайсосома строится поэтапно. Во-первых, SNRNP U1 связывает 5’-сайт сплайсинга, а SNRNP U2 связывает сайт ответвления.
Рисунок \(\PageIndex{4}\). (CC BY-NC-SA; Агатман )
Ряд других мяРНП ( U4, U6 и U5 ) связывают транскрипт пре-мРНК, образуя зрелый сплайсосомный комплекс . Это приводит к тому, что интрон образует петлю и объединяет 5′-сайт сплайсинга и 3′-сайт сплайсинга.
Рисунок \(\PageIndex{5}\). (CC BY-NC-SA; Agathman )
Теперь, когда сплайсосома собрана, можно начинать сплайсинг. Сначала разрезают 5’-конец интрона. Затем 5′-конец GU интрона соединяется с сайтом разветвления A, что создает лариатную структуру.
Рисунок \(\PageIndex{6}\). (CC BY-NC-SA; Agathman )
На этой стадии высвобождаются мяРНП U1 и U4 и расщепляется 3’-сайт сплайсинга. После полного расщепления интрона два экзона присоединяются друг к другу. Интрон в форме лариата высвобождается вместе с мяРНП U2, U5 и U6.
Рисунок \(\PageIndex{7}\). (CC BY-NC-SA; Agathman )
Интрон будет расщеплен, и мяРНП снова будут использоваться для сплайсинга других пре-мРНК. Зрелый транскрипт мРНК теперь готов к экспорту в цитоплазму для трансляции.
Альтернативный сплайсинг
Пример гена с одним интроном и двумя экзонами, использованный выше, представляет собой очень простую модель сплайсинга РНК. Многие гены содержат несколько экзонов, а также несколько интронов. Процесс, известный как альтернативный сплайсинг , позволяет включать различные комбинации экзонов в окончательную зрелую мРНК, создавая разные версии белков (называемых изоформами ), которые все кодируются одним и тем же геном. Альтернативный сплайсинг мРНК позволяет получить множество белков с разными функциями, причем все они производятся из одного гена. Одним из наиболее ярких примеров альтернативного сплайсинга является ген Dscam в Drosophila melanogaster (дрозофила). Этот единственный ген содержит 116 экзонов! Некоторые экзоны всегда включены, другие могут быть включены или не включены. У
Альтернативный сплайсинг мРНК позволяет получить множество белков с разными функциями, причем все они производятся из одного гена. Одним из наиболее ярких примеров альтернативного сплайсинга является ген Dscam в Drosophila melanogaster (дрозофила). Этот единственный ген содержит 116 экзонов! Некоторые экзоны всегда включены, другие могут быть включены или не включены. У
Ниже приведен пример альтернативного сплайсинга транскрипта пре-мРНК. В этом случае из этой пре-мРНК можно получить две разные альтернативно сплайсированные мРНК. Две зрелые мРНК могут содержать либо желтый, либо зеленый экзон. Это дает две различные изоформы белка, когда мРНК транслируются в белок.
Учебное пособие по сплайсингу мРНК, подготовленное доктором Кэтрин Харрис.
Это руководство было профинансировано грантом Title V-STEM #P031S0

Эта страница под названием «Сплайсинг мРНК» публикуется в соответствии с лицензией CC BY-NC-SA, ее автором, ремиксом и/или куратором является Кэтрин Харрис.
- Наверх
- Была ли эта статья полезной?
- Тип изделия
- Раздел или Страница
- Автор
- Кэтрин Харрис
- Лицензия
- CC BY-NC-SA
- Показать оглавление
- нет
- Теги

Механизм альтернативного сплайсинга и его регуляция
1. Гилберт В. Почему гены разбиты на части? Природа. 1978; 271:501. дои: 10.1038/271501a0. [PubMed] [CrossRef] [Google Scholar]
2. C. elegans Sequencing Consortium, соавтор. Последовательность генома нематоды C. elegans: платформа для изучения биологии. Наука. 1998;282:2012–2018. doi: 10.1126/science.282.5396.2012. [PubMed] [CrossRef] [Google Scholar]
3. Консорциум секвенирования генома человека I Международный секвенатор генома человека C, соавтор. Завершение эухроматической последовательности генома человека. Природа. 2004; 431:931–945. doi: 10.1038/nature03001. [PubMed] [CrossRef] [Google Scholar]
4. Nilsen TW, Graveley BR. Расширение эукариотического протеома путем альтернативного сплайсинга. Природа. 2010; 463:457–463. doi: 10.1038/nature08909. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
5. Zheng CL, Fu XD, Gribskov M. Характеристики и регуляторные элементы, определяющие конститутивный сплайсинг и различные способы альтернативного сплайсинга у человека и мыши. РНК. 2005; 11: 1777–1787. doi: 10.1261/РНК.2660805. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Характеристики и регуляторные элементы, определяющие конститутивный сплайсинг и различные способы альтернативного сплайсинга у человека и мыши. РНК. 2005; 11: 1777–1787. doi: 10.1261/РНК.2660805. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
6. Effenberger KA, Perriman RJ, Bray WM, et al. Высокопроизводительный анализ сплайсинга идентифицирует новые классы ингибиторов сплайсосом человека и дрожжей. J Биомоль Экран. 2013;18:1110–1120. дои: 10.1177/1087057113493117. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
7. Wahl MC, Will CL, Luhrmann R. Сплайсосома: принципы проектирования динамической машины RNP. Клетка. 2009; 136: 701–718. doi: 10.1016/j.cell.2009.02.009. [PubMed] [CrossRef] [Google Scholar]
8. Ким Э., Горен А., Аст Г. Альтернативный сплайсинг: текущие перспективы. Биоэссе. 2008; 30:38–47. doi: 10.1002/bies.20692. [PubMed] [CrossRef] [Google Scholar]
9. Иримиа М., Пенни Д., Рой С.В. Коэволюция числа геномных интронов и сайтов сплайсинга. Тенденции Жене. 2007; 23: 321–325. doi: 10.1016/j.tig.2007.04.001. [PubMed] [CrossRef] [Академия Google]
Тенденции Жене. 2007; 23: 321–325. doi: 10.1016/j.tig.2007.04.001. [PubMed] [CrossRef] [Академия Google]
10. Fu G, Condon KC, Epton MJ, et al. Летальность насекомых, характерная для самок, сконструирована с использованием альтернативного сплайсинга. Нац биотехнолог. 2007; 25: 353–357. doi: 10.1038/nbt1283. [PubMed] [CrossRef] [Google Scholar]
11. Hurt KJ, Sezen SF, Champion HC, et al. Альтернативно, сплайсированная нейрональная синтаза оксида азота опосредует эрекцию полового члена. Proc Natl Acad Sci USA. 2006; 103:3440–3443. doi: 10.1073/pnas.0511326103. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
12. Blencowe BJ. Альтернативный сплайсинг: новые идеи глобального анализа. Клетка. 2006; 126:37–47. doi: 10.1016/j.cell.2006.06.023. [PubMed] [CrossRef] [Академия Google]
13. Nygard AB, Cirera S, Gilchrist MJ, et al. Изучение альтернативного сплайсинга у свиней. Примечания BMC Res. 2010;3:123. дои: 10.1186/1756-0500-3-123. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
14. Kelemen O, Convertini P, Zhang Z, et al. Функция альтернативного сплайсинга. Ген. 2013; 514:1–30. doi: 10.1016/j.gene.2012.07.083. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Kelemen O, Convertini P, Zhang Z, et al. Функция альтернативного сплайсинга. Ген. 2013; 514:1–30. doi: 10.1016/j.gene.2012.07.083. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
15. Kim E, Magen A, Ast G. Различные уровни альтернативного сплайсинга среди эукариот. Нуклеиновые Кислоты Res. 2007; 35: 125–131. дои: 10.1093/нар/гкл924. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
16. Galante PA, Sakabe NJ, Kirschbaum-Slager N, de Souza SJ. Обнаружение и оценка событий удержания интронов в транскриптоме человека. РНК. 2004; 10: 757–765. doi: 10.1261/РНК.5123504. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
17. Sakabe NJ, de Souza SJ. Особенности последовательности, ответственные за удержание интронов у человека. Геномика BMC. 2007; 8:59. дои: 10.1186/1471-2164-8-59. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
18. Гольдстром А.С., Гринлиф А.Л., Гарсия-Бланко М.А. Котранскрипционный сплайсинг пре-мессенджерных РНК: рассмотрение механизма альтернативного сплайсинга. Ген. 2001; 277:31–47. doi: 10.1016/S0378-1119(01)00695-3. [PubMed] [CrossRef] [Google Scholar]
Ген. 2001; 277:31–47. doi: 10.1016/S0378-1119(01)00695-3. [PubMed] [CrossRef] [Google Scholar]
19. Di Giammartino DC, Nishida K, Manley JL. Механизмы и последствия альтернативного полиаденилирования. Мол Ячейка. 2011;43:853–866. doi: 10.1016/j.molcel.2011.08.017. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
20. Мастранжело А.М., Мароне Д., Лайдо Г. и др. Альтернативный сплайсинг: повышение способности справляться со стрессом за счет пластичности транскриптома. Растениевод. 2012; 185–186: 40–49. doi: 10.1016/j.plantsci.2011.09.006. [PubMed] [CrossRef] [Google Scholar]
21. Хоскинс А.А., Мур М.Дж. Сплайсосома: гибкая обратимая макромолекулярная машина. Тенденции биохимических наук. 2012; 37: 179–188. doi: 10.1016/j.tibs.2012.02.009. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
22. Wang Z, Burge CB. Регулирование сплайсинга: от перечня элементов регулирования до интегрированного кода сплайсинга. РНК. 2008; 14:802–813. doi: 10. 1261/rna.876308. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
1261/rna.876308. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
23. Ван З., Сяо Х., Ван Ностранд Э., Бердж С.Б. Общие и специфические функции экзонических глушителей сплайсинга в контроле сплайсинга. Мол Ячейка. 2006; 23:61–70. doi: 10.1016/j.molcel.2006.05.018. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
24. Jin Y, Yang Y, Zhang P. Новое понимание вторичной структуры РНК при альтернативном сплайсинге пре-мРНК. РНК биол. 2011; 8: 450–457. doi: 10.4161/rna.8.3.15388. [PubMed] [CrossRef] [Google Scholar]
25. Liu W, Zhou Y, Hu Z, et al. Регуляция активности энхансеров сплайсинга вторичными структурами РНК. ФЭБС лат. 2010; 584:4401–4407. doi: 10.1016/j.febslet.2010.090,039. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
26. McManus CJ, Graveley BR. Структура РНК и механизмы альтернативного сплайсинга. Curr Opin Genet Dev. 2011;21:373–379. doi: 10.1016/j.gde.2011.04.001. [Статья бесплатно PMC] [PubMed] [CrossRef] [Google Scholar]
27. Nasim FU, Hutchison S, Cordeau M, Chabot B. Высокоаффинные сайты связывания hnRNP A1 и инвертированные повторы, образующие дуплекс, оказывают сходное действие на 5′ выбор сайта сплайсинга в поддержку общего механизма образования петель и репрессии. РНК. 2002; 8: 1078–1089.. doi: 10.1017/S1355838202024056. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
Nasim FU, Hutchison S, Cordeau M, Chabot B. Высокоаффинные сайты связывания hnRNP A1 и инвертированные повторы, образующие дуплекс, оказывают сходное действие на 5′ выбор сайта сплайсинга в поддержку общего механизма образования петель и репрессии. РНК. 2002; 8: 1078–1089.. doi: 10.1017/S1355838202024056. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
28. Han SP, Kassahn KS, Skarshewski A, Ragan MA, Rothnagel JA, Smith R. Функциональные последствия появления альтернативного сплайсинга в hnRNP A/B стенограммы. РНК. 2010;16:1760–1768. doi: 10.1261/РНК.2142810. [Статья бесплатно PMC] [PubMed] [CrossRef] [Google Scholar]
29. Wang E, Cambi F. Гетерогенные ядерные рибонуклеопротеины H и F регулируют соотношение протеолипидный белок/DM20, рекрутируя малый ядерный рибонуклеопротеин U1 через сложную группу G. бежит. Дж. Биол. Хим. 2009 г.;284:11194–11204. doi: 10.1074/jbc.M809373200. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
30. Park E, Iaccarino C, Lee J, et al. Регуляторные роли гетерогенного ядерного рибонуклеопротеина М и белка Nova-1 в альтернативном сплайсинге пре-мРНК дофаминового рецептора D2. Дж. Биол. Хим. 2011; 286:25301–25308. doi: 10.1074/jbc.M110.206540. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Регуляторные роли гетерогенного ядерного рибонуклеопротеина М и белка Nova-1 в альтернативном сплайсинге пре-мРНК дофаминового рецептора D2. Дж. Биол. Хим. 2011; 286:25301–25308. doi: 10.1074/jbc.M110.206540. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
31. Liu G, Razanau A, Hai Y, et al. Консервативный серин гетерогенного ядерного рибонуклеопротеина L (hnRNP L) опосредует регулируемый деполяризацией альтернативный сплайсинг калиевых каналов. Дж. Биол. Хим. 2012;287:22709–22716. doi: 10.1074/jbc.M112.357343. [Статья PMC free] [PubMed] [CrossRef] [Google Scholar]
32. Yu J, Hai Y, Liu G, Fang T, Kung SK, Xie J. Гетерогенный ядерный рибонуклеопротеин L является важным компонентом Ca2+/ регулируемый кальмодулин-зависимой протеинкиназой IV альтернативный сплайсинг через цитидин-аденозиновые повторы. Дж. Биол. Хим. 2009; 284:1505–1513. doi: 10.1074/jbc.M805113200. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
33. Ghosh G, Adams JA. Механизм фосфорилирования и структура серин-аргининовых протеинкиназ.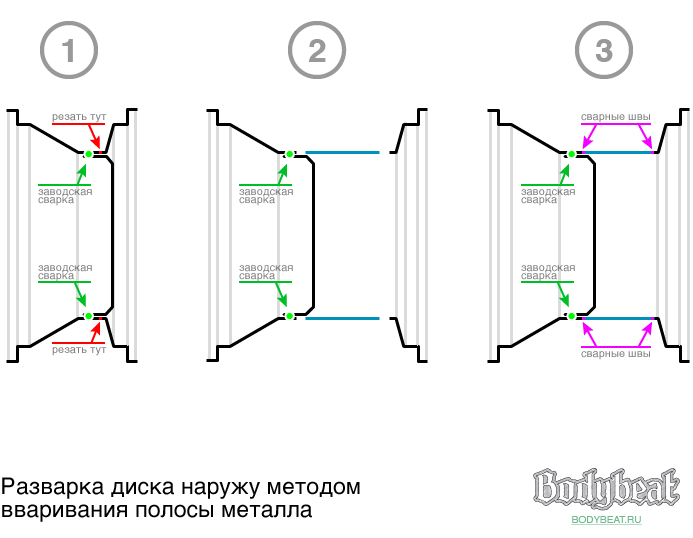 FEBS J. 2011; 278: 587–59.7. doi: 10.1111/j.1742-4658.2010.07992.x. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
FEBS J. 2011; 278: 587–59.7. doi: 10.1111/j.1742-4658.2010.07992.x. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
34. Twyffels L, Gueydan C, Kruys V. Челночные SR-белки: больше, чем факторы сплайсинга. FEBS J. 2011; 278:3246–3255. doi: 10.1111/j.1742-4658.2011.08274.x. [PubMed] [CrossRef] [Google Scholar]
35. Лонг Дж. К., Касерес Дж. Ф. Семейство белков SR факторов сплайсинга: основные регуляторы экспрессии генов. Биохим Дж. 2009; 417:15–27. doi: 10.1042/BJ20081501. [PubMed] [CrossRef] [Академия Google]
36. Калнина З., Заякин П., Силина К., Лине А. Изменения сплайсинга пре-мРНК при раке. Гены Хромосомы Рак. 2005; 42: 342–357. doi: 10.1002/gcc.20156. [PubMed] [CrossRef] [Google Scholar]
37. Beyer AL, Bouton AH, Miller OL., Jr Корреляция структуры hnRNP и расщепления зарождающегося транскрипта. Клетка. 1981; 26: 155–165. doi: 10.1016/0092-8674(81)
-3. [PubMed] [CrossRef] [Google Scholar]
38. Kornblihtt AR, de la Mata M, Feeda JP, et al. Множественные связи между транскрипцией и сплайсингом. РНК. 2004;10:1489–1498. doi: 10.1261/РНК.7100104. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Множественные связи между транскрипцией и сплайсингом. РНК. 2004;10:1489–1498. doi: 10.1261/РНК.7100104. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
39. Монтес М., Бесерра С., Санчес-Альварес М., Суне С. Функциональное сочетание транскрипции и сплайсинга. Ген. 2012; 501:104–117. doi: 10.1016/j.gene.2012.04.006. [PubMed] [CrossRef] [Google Scholar]
40. Ryman K, Fong N, Bratt E, Bentley DL, Ohman M. С-концевой домен РНК Pol II помогает гарантировать, что редактирование предшествует сплайсингу транскрипта GluR-B. . РНК. 2007; 13:1071–1078. doi: 10.1261/rna.404407. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
41. Бентли Д.Л. Правила взаимодействия: ко-транскрипционное рекрутирование факторов процессинга пре-мРНК. Curr Opin Cell Biol. 2005; 17: 251–256. doi: 10.1016/j.ceb.2005.04.006. [PubMed] [CrossRef] [Google Scholar]
42. Bartkowiak B, Liu P, Phatnani HP, et al. CDK12 представляет собой киназу CTD, связанную с удлинением транскрипции, ортолог многоклеточных животных дрожжевого Ctk1. Гены Дев. 2010;24:2303–2316. doi: 10.1101/gad.1968210. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Гены Дев. 2010;24:2303–2316. doi: 10.1101/gad.1968210. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
43. Kim H, Erickson B, Luo W, et al. Фосфорилирование генспецифической РНК-полимеразы II и код CTD. Nat Struct Mol Biol. 2010;17:1279–1286. doi: 10.1038/nsmb.1913. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
44. Phatnani HP, Greenleaf AL. Фосфорилирование и функции РНК-полимеразы II CTD. Гены Дев. 2006; 20: 2922–2936. doi: 10.1101/gad.1477006. [PubMed] [CrossRef] [Google Scholar]
45. Розонина Э., Бленкоу Б.Дж. Анализ потребности в гептапептидных повторах CTD РНК-полимеразы II при сплайсинге пре-мРНК и расщеплении 3′-конца. РНК. 2004; 10: 581–589. doi: 10.1261/РНК.5207204. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
46. Калсотра А., Купер Т.А. Функциональные последствия регулируемого развитием альтернативного сплайсинга. Нат Рев Жене. 2011;12:715–729. doi: 10.1038/nrg3052. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
47. Schor IE, Gómez Acuña LI, Kornblihtt AR. Связь между транскрипцией и альтернативным сплайсингом. Лечение рака Res. 2013; 158:1–24. [PubMed] [Google Scholar]
Schor IE, Gómez Acuña LI, Kornblihtt AR. Связь между транскрипцией и альтернативным сплайсингом. Лечение рака Res. 2013; 158:1–24. [PubMed] [Google Scholar]
48. Dujardin G, Lafaille C, Petrillo E, et al. Транскрипционная элонгация и альтернативный сплайсинг. Биохим Биофиз Акта. 2013;1829: 134–140. doi: 10.1016/j.bbagrm.2012.08.005. [PubMed] [CrossRef] [Google Scholar]
49. Gómez Acuña LI, Fiszbein A, Alló M, et al. Связи между хроматиновыми сигнатурами и сплайсингом. Wiley Interdiscip Rev RNA. 2013; 4:77–91. doi: 10.1002/wrna.1142. [PubMed] [CrossRef] [Google Scholar]
50. Iannone C, Valcárcel J. Нить Хроматина к альтернативному регулированию сплайсинга. Хромосома. 2013; 122:465–474. doi: 10.1007/s00412-013-0425-x. [PubMed] [CrossRef] [Академия Google]
51. Шукла С., Обердорфер С. Котранскрипционная регуляция альтернативного сплайсинга пре-мРНК. Биохим Биофиз Акта. 2012; 1819: 673–683. doi: 10.1016/j.bbagrm.2012.01.014. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
52. Ma X, Li-Ling J, Huang Q, et al. Систематический анализ альтернативных промоторов, коррелирующих с альтернативным сплайсингом в генах человека. Геномика. 2009;93:420–425. doi: 10.1016/j.ygeno.2009.01.008. [PubMed] [CrossRef] [Google Scholar]
Ma X, Li-Ling J, Huang Q, et al. Систематический анализ альтернативных промоторов, коррелирующих с альтернативным сплайсингом в генах человека. Геномика. 2009;93:420–425. doi: 10.1016/j.ygeno.2009.01.008. [PubMed] [CrossRef] [Google Scholar]
53. Xin D, Hu L, Kong X. Альтернативные промоторы влияют на альтернативный сплайсинг на геномном уровне. ПЛОС Один. 2008;3:e2377. doi: 10.1371/journal.pone.0002377. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
54. Шабалина С.А., Спиридонов А.Н., Спиридонов Н.А., Кунин Е.В. Связи между альтернативной транскрипцией и альтернативным сплайсингом у млекопитающих. Геном Биол Эвол. 2010;2:791–799. doi: 10.1093/gbe/evq058. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
55. Рой Б., Хаупт Л.М., Гриффитс Л.Р. Обзор: Альтернативный сплайсинг (АС) генов как подход к созданию сложных белков. Карр Геномикс. 2013;14:182–194. doi: 10.2174/1389202911314030004. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
56. Ларо Л.Ф., Брукс А.Н., Сергель Д.А., Менг К., Бреннер С.Е. Сочетание альтернативного сплайсинга и нонсенс-опосредованного распада мРНК. Adv Exp Med Biol. 2007; 623:190–211. [PubMed] [Google Scholar]
Ларо Л.Ф., Брукс А.Н., Сергель Д.А., Менг К., Бреннер С.Е. Сочетание альтернативного сплайсинга и нонсенс-опосредованного распада мРНК. Adv Exp Med Biol. 2007; 623:190–211. [PubMed] [Google Scholar]
57. Льюис Б.П., Грин Р.Е., Бреннер С.Е. Доказательства широко распространенного сочетания альтернативного сплайсинга и нонсенс-опосредованного распада мРНК у людей. Proc Natl Acad Sci USA. 2003; 100:189–192. doi: 10.1073/pnas.0136770100. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
58. Nyiko T, Kerenyi F, Szabadkai L, et al. Нонсенс-опосредованный распад мРНК растений контролируется различными ауторегуляторными цепями и может индуцироваться EJC-подобным комплексом. Нуклеиновые Кислоты Res. 2013;41:6715–6728. дои: 10.1093/нар/гкт366. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
59. Kalyna M, Simpson CG, Syed NH, et al. Альтернативный сплайсинг и нонсенс-опосредованный распад модулируют экспрессию важных регуляторных генов у арабидопсиса. Нуклеиновые Кислоты Res. 2012;40:2454–2469. doi: 10.1093/nar/gkr932. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Нуклеиновые Кислоты Res. 2012;40:2454–2469. doi: 10.1093/nar/gkr932. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
60. Saltzman AL, Kim YK, Pan Q, et al. Регуляция множественных ядерных сплайсосомных белков путем нонсенс-опосредованного распада мРНК, связанного с альтернативным сплайсингом. Мол Селл Биол. 2008; 28:4320–4330. doi: 10.1128/MCB.00361-08. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
61. Ge Y, Porse BT. Функциональные последствия удержания интрона: альтернативный сплайсинг в сочетании с NMD как регулятор экспрессии генов. Биоэссе. 2014; 36: 236–243. doi: 10.1002/bies.201300156. [PubMed] [CrossRef] [Google Scholar]
62. Herai RH, Yamagishi ME. Обнаружение межхромосомного транс-сплайсинга человека в банках данных последовательностей. Кратко Биоинформ. 2010;11:198–209. doi: 10.1093/bib/bbp041. [PubMed] [CrossRef] [Google Scholar]
63. Horiuchi T, Aigaki T. Альтернативный транс-сплайсинг: новый способ процессинга пре-мРНК. Биол Клетка. 2006;98:135–140. doi: 10.1042/BC20050002. [PubMed] [CrossRef] [Google Scholar]
Биол Клетка. 2006;98:135–140. doi: 10.1042/BC20050002. [PubMed] [CrossRef] [Google Scholar]
64. Lee SW, Jeong JS. Использование транс-сплайсингового рибозима, нацеленного на опухоль, для лечения рака. Методы Мол Биол. 2014;1103:83–95. [PubMed] [Google Scholar]
65. Jung HS, Lee SW. Рибозим-опосредованное избирательное уничтожение раковых клеток, экспрессирующих РНК карциноэмбрионального антигена, путем направленного транс-сплайсинга. Biochem Biophys Res Commun. 2006; 349: 556–563. doi: 10.1016/j.bbrc.2006.08.073. [PubMed] [CrossRef] [Академия Google]
66. Авале М.Е., Родригес-Мартин Т., Галло Дж.М. Транс-сплайсинговая коррекция дисбаланса изоформ тау в мышиной модели неправильного сплайсинга тау. Хум Мол Жене. 2013;22:2603–2611. doi: 10.1093/hmg/ddt108. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
67. Луко Р.Ф., Мистели Т. Больше, чем код сплайсинга: интеграция роли РНК, хроматина и некодирующей РНК в регуляции альтернативного сплайсинга. Curr Opin Genet Dev. 2011;21:366–372. doi: 10.1016/j.gde.2011.03.004. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Curr Opin Genet Dev. 2011;21:366–372. doi: 10.1016/j.gde.2011.03.004. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
68. Сингх Р.К., Купер Т.А. Сплайсинг пре-мРНК при заболеваниях и терапии. Тренды Мол Мед. 2012; 18: 472–482. doi: 10.1016/j.molmed.2012.06.006. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
69. Zhang J, Manley JL. Нарушение регуляции альтернативного сплайсинга пре-мРНК при раке. Рак Дисков. 2013;3:1228–1237. doi: 10.1158/2159-8290.CD-13-0253. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
70. Shkreta L, Bell B, Revil T, et al. Связанные с раком нарушения в альтернативном сплайсинге пре-мессенджерной РНК. Лечение рака Res. 2013; 158:41–94. [PubMed] [Google Scholar]
71. Kim YJ, Kim HS. Альтернативный сплайсинг и его роль в качестве диагностического маркера рака. Геномика Информ. 2012;10:74–80. doi: 10.5808/GI.2012.10.2.74. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
72. Лапук А.В., Волик С.В., Ван Ю, Коллинз С.С. Роль сплайсинга мРНК при раке предстательной железы. Азиат Джей Андрол. 2014;16:515–521. doi: 10.4103/1008-682X.127825. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Лапук А.В., Волик С.В., Ван Ю, Коллинз С.С. Роль сплайсинга мРНК при раке предстательной железы. Азиат Джей Андрол. 2014;16:515–521. doi: 10.4103/1008-682X.127825. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
73. Pajares MJ, Ezponda T, Catena R, et al. Альтернативный сплайсинг: новая тема в молекулярной и клинической онкологии. Ланцет Онкол. 2007;8:349–357. doi: 10.1016/S1470-2045(07)70104-3. [PubMed] [CrossRef] [Google Scholar]
74. Sitohy B, Nagy JA, Dvorak HF. Анти-VEGF/VEGFR терапия рака: переоценка мишени. Рак Рез. 2012;72:1909–1914. doi: 10.1158/0008-5472.CAN-11-3406. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
75. Pal S, Gupta R, Davuluri RV. Альтернативная транскрипция и альтернативный сплайсинг при раке. Фармакол Тер. 2012; 136: 283–294. doi: 10.1016/j.pharmthera.2012.08.005. [PubMed] [CrossRef] [Академия Google]
76. Pan Q, Shai O, Lee LJ, et al. Глубокое исследование сложности альтернативного сплайсинга в транскриптоме человека с помощью высокопроизводительного секвенирования.


 После этого остудите и приступайте к приготовлению суши.
После этого остудите и приступайте к приготовлению суши.